虚拟环境简介
单细胞转录组测序仿真及数据分析系统为东南大学生物科学与医学工程学院定制,服务于本科生物数据分析实战训练任务。目前预发布仿真实验模块。
仿真实验地点、仪器和试剂具体信息未在文档中公开。

实验原理
细胞悬液制备
组织样本先通过物理方式剪碎,使其能够充分与胰酶溶液接触。胰酶溶液消化细胞,降解细胞间结合处蛋白,迫使细胞分离,最终稀释处理为单细胞悬液。单细胞悬液制备的目的在于令各个细胞通入微流控芯片时不产生重叠交错的现象,最终一种条形码仅对应于单个细胞。
细胞标记
该步骤主要有两个目的:一为在体系中加入后续细胞裂解与mRNA逆转录过程中需要使用的酶与缓冲液;二是通过微流控芯片的“双十字”结构形成油包水液滴,使连有条形码的凝胶珠与单细胞被封装于同一液滴中。凝胶珠的浓度应大于细胞浓度,确保一颗凝胶珠不与一个以上的细胞共同包裹。该步骤原理图如下:
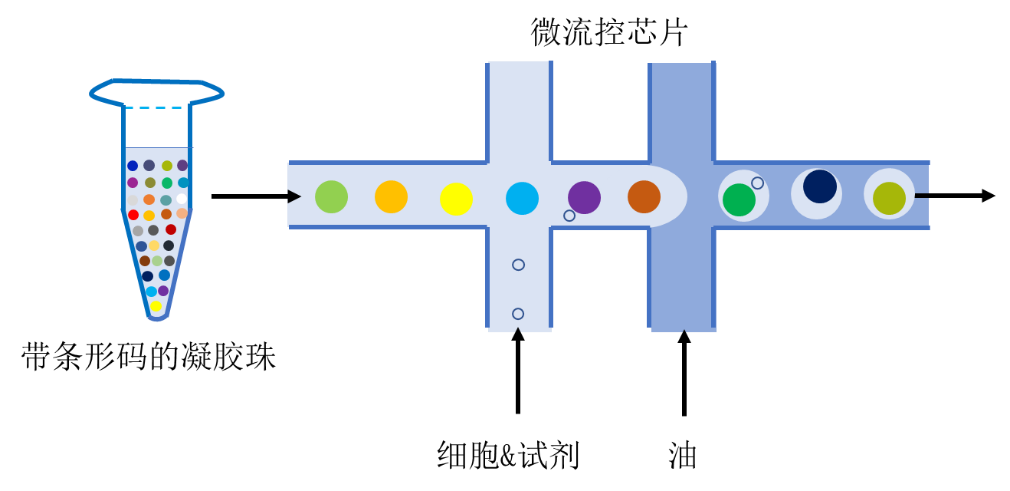
cDNA一链获取
经过细胞标记,有效封装的液滴内含有细胞、凝胶珠、TSO、细胞裂解液与逆转录体系。当封装完成后,裂解液开始反应,并于数秒后完成细胞裂解,并释放出mRNA。凝胶珠所连接的引物序列包括了以下几个部分:Illumina Read 1 测序引物、16 nt条形码序列(每个凝胶珠的条形码序列不相同,用于区分不同细胞)、12 nt unique molecular identifier (UMI)(区分同一细胞的不同转录本并去除PCR Duplications,实现绝对定量)、30 nt poly dT反转录引物。由于引物中包含30 nt poly dT反转录引物,可抓取带有polyA的mRNA。在逆转录加热过程中,凝胶珠自动溶解,释放出大量引物并反转录为cDNA一链,进而以SMART方式进行后续合成。当逆转录酶作用延伸至mRNA 5’ 末端真核mRNA特有的帽子结构时,会在末端连续添加几个胞嘧啶核苷酸。TSO末端的鸟嘌呤核苷酸与之互补配对,在逆转录酶的模板转换活性下以TSO为模板完成延伸。该步原理图如下:
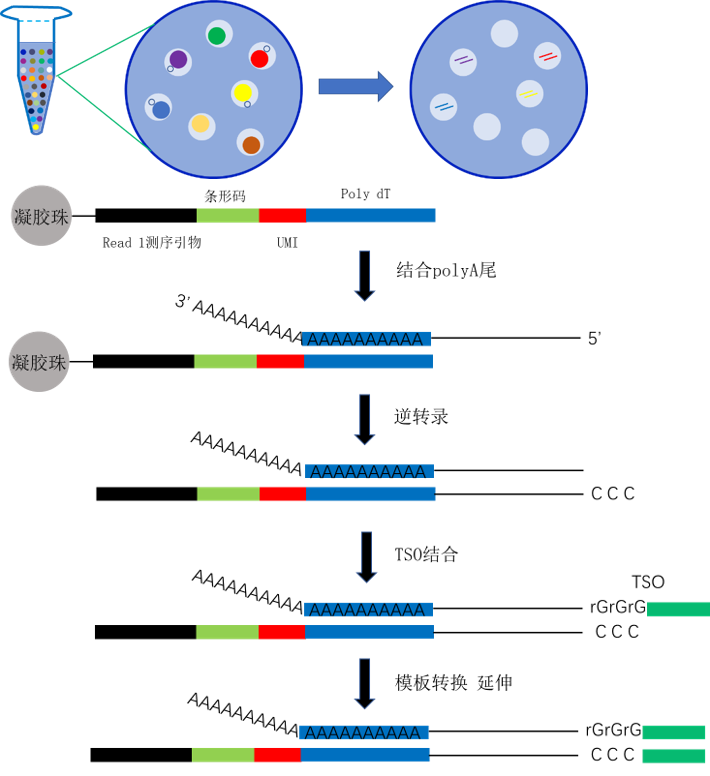
cDNA 富集
该步骤主要包括了液滴破乳、磁珠纯化cDNA与cDNA扩增。由于cDNA一链已带有条形码和UMI用以区分各个分子,可破乳合并来自于各个细胞的cDNA并进行常规PCR扩增。该步骤示意图如下:
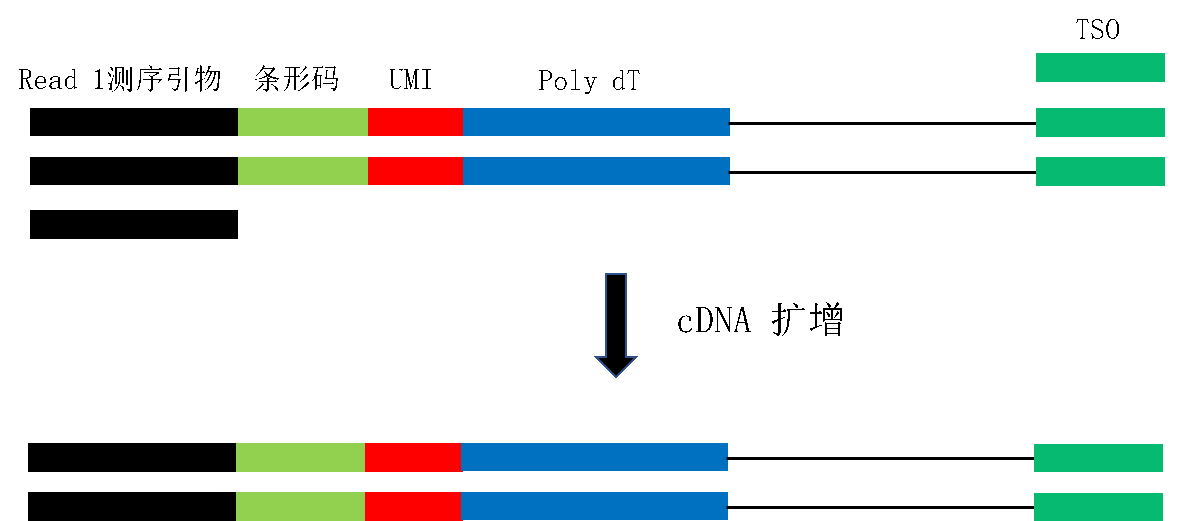
文库构建
该步骤为常规测序文库制备过程,使样本连接P5、P7端测序接头,其中P7端含有区别样本的index。该步骤流程如下图所示:
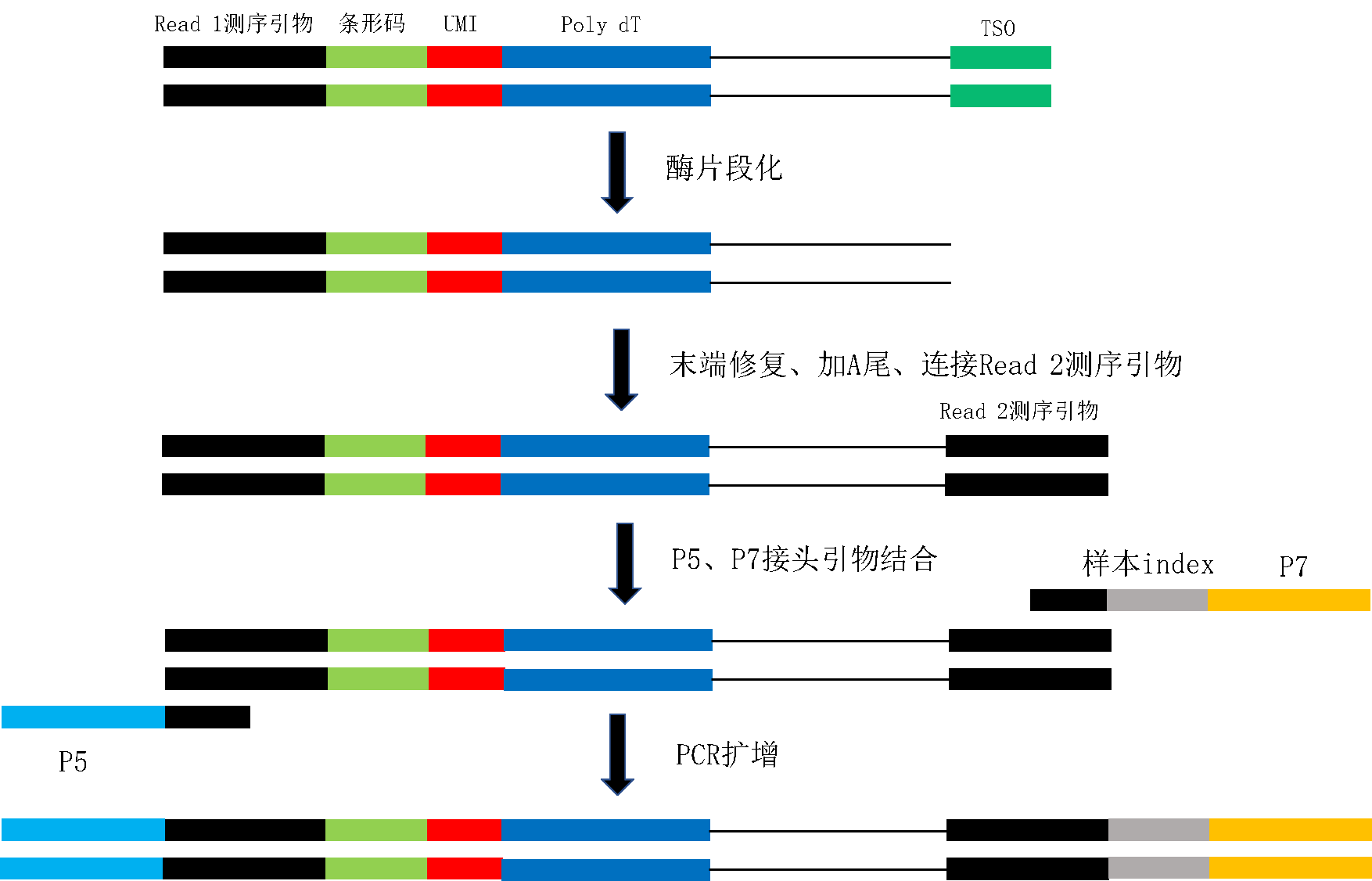
实验步骤
一、 细胞悬液制备
1) 用剪刀将组织剪成1~2 mm3的碎块,放入离心管内。
2) 加入胰酶溶液1~2 mL,37℃孵育30 min,期间不时震荡或吹打。
3) 收集细胞悬液,以300目尼龙网过滤,除去剩余组织,离心去除细胞碎片。
4) 加入2 mL PBS缓冲液充分混匀,4℃,1500 rpm/min离心弃上清,加入0.5 mL PBS缓冲液重悬细胞。
5) 细胞计数,稀释浓度至单细胞水平。
二、 细胞标记
1) 配制含逆转录酶、逆转录缓冲液、模板转换寡核苷酸(TSO)的混合液;冰上将细胞悬液与上述混合液混合,缓慢吹吸混匀。
2) 将凝胶珠涡旋30 s,使其均匀分散在含有裂解液的缓冲液中,微离心去除气泡。
3) 冰上将50 µl凝胶珠悬液、70 µl含细胞和酶的混合液、45 µl油通入微流控芯片,流速分别设为5、5、35 μL/h,收集液滴于200 μL PCR管内。
三、 cDNA制备与富集
A. 逆转录PCR
将PCR管放置于PCR仪内,设置逆转录程序如下:
| Step | Temperature | Time |
|---|---|---|
| 1 | 53°C | 45 min |
| 2 | 85°C | 5 min |
| 3 | 4°C | Hold |
结束后,转移样品至1.5 mL离心管中,向样品中加入125 μL破乳剂,静置2 min分层,缓慢用移液枪吸走底部油相。
B. 磁珠纯化
1) 在样品中加入200 μL磁珠,吸打混匀,室温孵育10 min。
2) 微离心,将溶液放置在磁力架上3min或者等到溶液变的清亮后,去除上清。
3) 在离心管中加入200 μL的新鲜的80%的酒精,孵育30 s,去除上清。
4) 重复上一步骤。
5) 为了彻底去除管中的酒精,用小量程的枪去除上清,放置1 min使残余酒精挥发。
6) 从磁力架上取下离心管,加入35.5 μL的洗脱液,吹吸混匀,室温孵育2 min。
7) 将离心管放在磁力架上超过1min,等到溶液清亮后,吸取35 μL上清放在一个新的200 μL离心管内。
C. cDNA扩增
配制65 μL cDNA扩增反应混合液(50 μL扩增预混液、15 μL cDNA引物),吹吸混匀。将该混合液加入样品中,设置PCR程序:
| Step | Temperature | Time |
|---|---|---|
| Initial Denaturation | 98℃ | 3 min |
| Denaturation | 98℃ | 15 sec |
| Annealing | 63℃ | 20 sec |
| Extension | 72℃ | 1 min |
| Final Extension | 72℃ | 1 min |
| Hold | 4℃ | Hold |
D. 磁珠二次纯化
1) 在样品中加入60 μL磁珠,吸打混匀,室温孵育5 min。
2) 微离心,将溶液放置在磁力架上3min或者等到溶液变的清亮后,去除上清。
3) 在离心管中加入200 μL的新鲜的80%的酒精,孵育30 s,去除上清。
4) 重复上一步骤。
5) 为了彻底去除管中的酒精,用小量程的枪去除上清,放置2 min使残余酒精挥发。
6) 从磁力架上取下离心管,加入40.5 μL的洗脱液,吹吸混匀,室温孵育2 min。
7) 将离心管放在磁力架上超过1min,等到溶液清亮后,吸取40 μL上清放在一个新的200 μL离心管内。
五、 文库构建
A. 酶切片段化、末端修复与加A尾
取10 μL样品转移至新的PCR管中,加入10 μL片段化酶、5 μL 片段化缓冲液、25 μL预混液(含末端补齐酶与DNA聚合酶),体系共50 μL,在冰上吹吸混匀,微离心,转移至PCR仪中预冷至4℃:
| Step | Temperature | Time |
|---|---|---|
| Pre-cool block | 4 °C | Hold |
| Fragmentation | 32 °C | 5 min |
| End Repair & A-tailing | 65 ℃ | 30 min |
| Hold | 4 °C | Hold |
B. 片段长度筛选
1) 在样品中加入30 μL磁珠,吸打混匀,室温孵育5 min。
2) 微离心,将溶液放置在磁力架上3min或者等到溶液变的清亮后,吸取75 μL上清转移到一个新的离心管中。
3) 在上清中加入10 μL磁珠,上下吸打,使得DNA与磁珠充分混匀。室温放置5 min。
4) 微离心,将溶液放置在磁力架上3min或者等到溶液变的清亮后,去除上清。
5) 在离心管中加入200 μL的新鲜的80%的酒精,孵育30s,去除上清。
6) 重复上一步骤。
7) 为了彻底去除管中的酒精,用小量程的枪去除上清。
8) 保持管子在磁力架上,打开盖子,室温下放置2 min。
9) 将离心管取下,加入50.5 μL 洗脱液,吸打混匀,涡旋10s,微离心。
10) 室温孵育2 min。
11) 将离心管放在磁力架上超过1min,等到溶液清亮后,吸取50 μL放在一个新的200 μL离心管内。
C. 引物接头连接
在样品中加入20 μL缓冲液、10 μL DNA连接酶、20 μL read2测序引物接头溶液,吹吸混匀,微离心,置于PCR仪中,热盖温度为30℃,反应总体系50μL,程序如下:
| Step | Temperature | Time |
|---|---|---|
| 1 | 20 °C | 15 min |
| 2 | 4°C | Hold |
D. 磁珠纯化
1) 在样品中加入80 μL磁珠,吸打混匀,室温孵育5 min。
2) 微离心,将溶液放置在磁力架上3min或者等到溶液变的清亮后,去除上清。
3) 在离心管中加入200 μL的新鲜的80%的酒精,孵育30 s,去除上清。
4) 重复上一步骤。
5) 为了彻底去除管中的酒精,用小量程的枪去除上清,放置2 min使残余酒精挥发。
6) 从磁力架上取下离心管,加入30.5 μL的洗脱液,吹吸混匀,室温孵育2 min。
7) 将离心管放在磁力架上超过1min,等到溶液清亮后,吸取30 μL上清放在一个新的200 μL离心管内。
E. index接头连接(P7端)
在样品中加入50 μL PCR预混液、10 μL index引物(P7端引物)和10 μL P5端引物,吹吸混匀,微离心,放入PCR仪中,程序如下:
| Step | Temperature | Time |
|---|---|---|
| Initial Denaturation | 98℃ | 45 sec |
| Denaturation | 98℃ | 20 sec |
| Annealing | 54℃ | 30 sec |
| Extension | 72℃ | 20 sec |
| Final Extension | 72℃ | 1 min |
| Hold | 4℃ | Hold |
F. 磁珠筛选片段大小
1) 在样品中加入60 μL磁珠,吸打混匀,室温孵育5 min。
2) 微离心,将溶液放置在磁力架上3min或者等到溶液变的清亮后,吸取150 μL上清转移到一个新的离心管中。
3) 在上清中加入20 μL磁珠,上下吸打,使得DNA与磁珠充分混匀。室温放置5 min。
4) 微离心,将溶液放置在磁力架上3min或者等到溶液变的清亮后,去除上清。
5) 在离心管中加入200 μL的新鲜的80%的酒精,孵育30s,去除上清。
6) 重复上一步骤。
7) 为了彻底去除管中的酒精,用小量程的枪去除上清。
8) 保持管子在磁力架上,打开盖子,室温下放置2 min。
9) 将离心管取下,加入35.5 μL 洗脱液,吸打混匀,涡旋10s,微离心。
10) 室温孵育2 min。
11) 将离心管放在磁力架上超过1min,等到溶液清亮后,吸取35 μL放在一个新的200 μL离心管内。
测试结果
于2020年4月8日12:30起,连续进行三次测试,每次测试进行一次完整的单细胞测序实验仿真。测试环境基本信息如下:
| 名称 | 指标 |
|---|---|
| 操作系统 | Windows 10 x64 企业版 LTSC |
| 处理器 | Intel® Core™ i7-8750H CPU @ 2.20 GHz 2.21GHz |
| 内存 | 16.0GB |
| 分辨率 | 1920 * 1080 |
资源占用
平均内存占用219M,CPU占用39.9%(12线程皆有占用)。高CPU占用导致测试设备过热,局部达80℃以上。
用户体验
测试环境下操作基本无卡顿。
软件启动默认全屏,无正常退出方法;分辨率须手动调整,无法完全适应测试环境显示。
第一视角无法位移,只可按ctrl缩放距离,为部分操作带来不便。部分对象灵敏度欠佳,须多次点击才可触发事件。
重要漏洞
- 第三次测试的第20分40秒,移液枪对象归位异常,保持随鼠标移动,致使后续步骤无法选择该对象进行操作;
- 三次测试中,第4次和第5次PCR后热循环仪打开,无可选择对象,实验仿真无法继续。目前连续点击热循环仪运行按钮两次似乎能继续实验。
仿真性
- 大部分离心操作缺乏配平步骤;
- 移液枪和枪头种类单一,依实验步骤来看应至少准备
2~20μL和20~200μL两种型号; - PCR反应的循环步骤设定皆被忽略,不符合真实情况。例如第2次PCR反应,
Denaturation、Annealing、Extension三个步骤应执行12次循环
安全性
仿真实验、测验的数据文件以txt和json格式暴露在主文件夹Single Cell ATAC_Data\StreamingAssets中,易被篡改或误改,进而影响使用。
建议
建议依照测试报告所列问题逐条改进。
- 优先级:重要漏洞>安全性>仿真性>用户体验>资源占用
- 用户体验部分不妨参考市面上流行的第一人称PC游戏,尤其是沙盒类。
- 安全性方面,可考虑应用由桌面迁移至web端,数据入库。